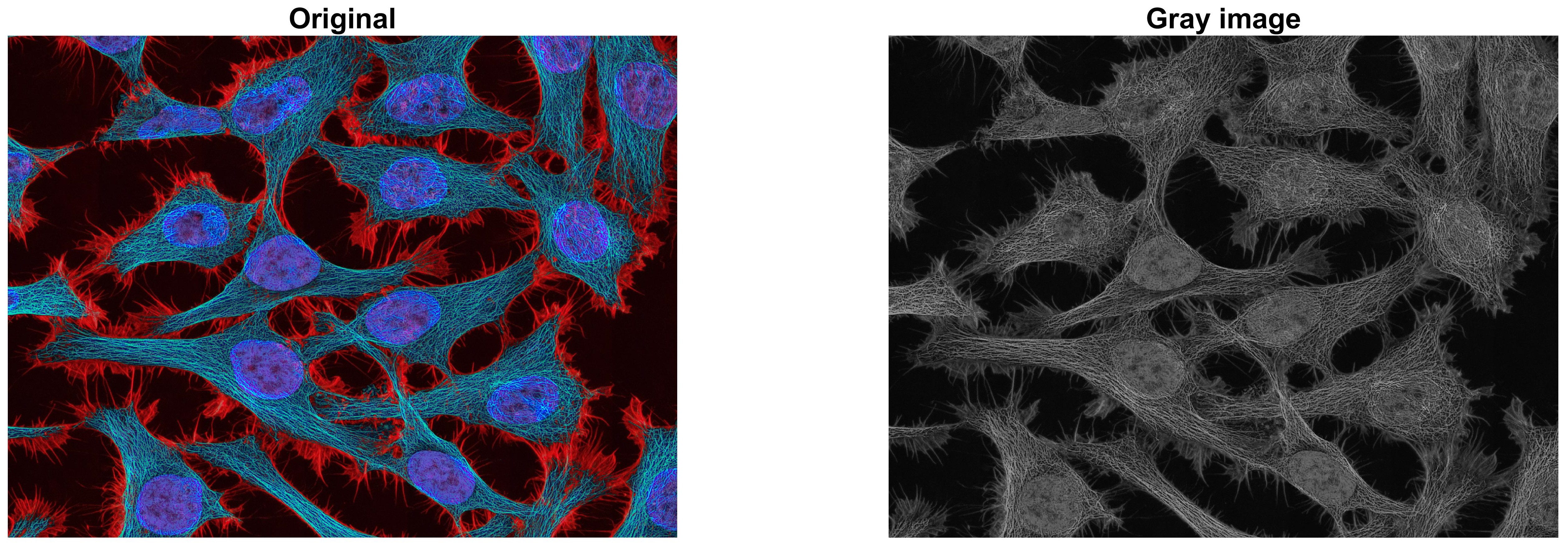
앞의 글 [Python 영상처리 기초 3] 이미지의 픽셀값 다루기 에서 BGR 컬러채널을 갖는
이미지의 gray scale 변환을 수행하였습니다.
그리고 gray scale 변환된 이미지 데이터는 기본적으로 uint8 형식을 가짐을 알 수 있었습니다.
Normalization
보통 Image Processing은 float (혹은 소숫점) 형식의 이미지 데이터를 다룹니다.
만일 정수형(int 또는 uint)의 픽셀값을 대상으로 사칙연산(곱셈, 나눗셈 등)을 한다면 연산후
소수점 이하의 값은 제거된다거나 표현가능 범위(여기서 uint8는 0 부터 255까지) 를
넘어서는 경우등 연산결과가 이미지상에 제대로 표현할 수 없기 때문입니다.
float 등의 소수점 형식의 이미지 데이터는 최소 0부터 최대 1.0 까지 값의 범위를 갖도록 정규화 하는것이
일반적 입니다. uint8로부터의 변환 방법은, 간단하게도, 데이터 타입 변환( array.astype('data type') ) 후
각 픽셀값을 (표현가능 범위)최대값인 255로 나누는 것입니다.
( 위 이미지는 본 블로그 맨밑 출처 1에 가셔서 다운로드 받을 수 있습니다. )
변환하는 코드 또한 간단합니다.
import numpy as np
img_gray = img_gray.astype(np.float32) # convert from uint8 to float32 type data
img_gray /= 255. # scale conversion from 0-255.0 to 0-1.0
위의 작업을 통해 이미지 데이터 "img_gray" 는 픽셀값의 형식만이 변환된 것이므로 픽셀값의 분포는
변하지 않았을 것입니다. 그리고 앞의 글 [Python 영상처 기초 3] 이미지의 픽셀값 다루기 - Histogram
에 표시된 histogram에서 보듯이 대상 이미지의 gray scale (uint8 형식) 최소값은 0 이 아니고 최대값
또한 255가 아니였습니다.
만일 이미지 데이터의 최소, 최대값을 알고 있다면 이를 이용해 픽셀값의 분포범위를 0 - 1.0로써
rescaling이 - 픽셀의 최소값이 0, 최대값이 1.0으로써 rescaling을 뜻함 - 가능하며 픽셀값 분포가 넓게
퍼짐으로써 이미지 품질개선의 효과를 가질 수 있습니다.
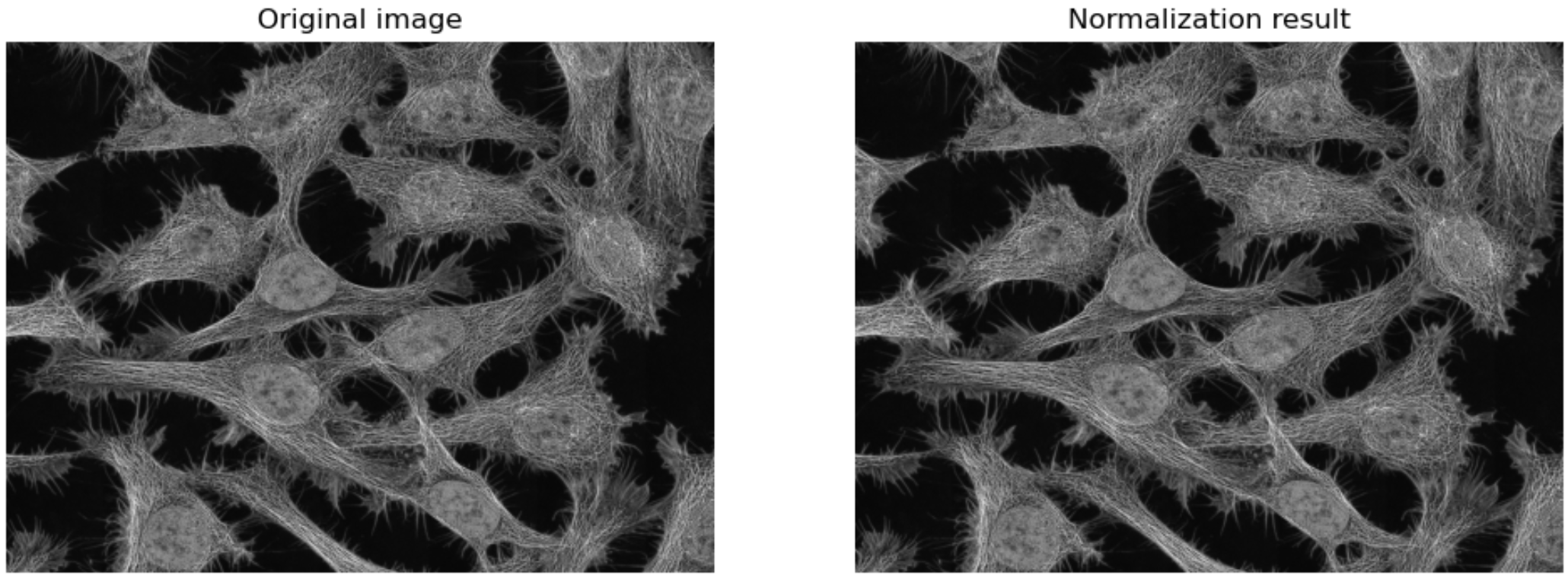
본 글에서는 간단하면서도 널리 사용되는 Min-Max Normalization을 수행해 보겠습니다.
(머신러닝에서 MNIST 등의 이미지를 다룰때에도 기본적으로 이용되는 테크닉 입니다.)
Min-Max Normalization의 방법은 대상 이미지의 픽셀값을 최소값으로 뺀후 최대, 최소값 차이로 나눕니다.
min-max scaling = (X - Xmin) / (Xmax - Xmin)
(여기서 X: 픽셀값, Xmax: 픽셀의 최대값, Xmin: 픽셀의 최소값)
주의할점은 입력 이미지 데이터는 소수점 형식 (float32 등)의 변수로 변환해야 하며
각 픽셀별로 나눗셈 연산을 수행하므로 연산 결과값 또한 소수점 형식의 변수에 저장되어야 합니다.
# data type conversion and rescaling
img_gray_copy = img_gray.copy().astype(np.float32) # convert from uint8 to float32 type data
img_gray_copy /= 255. # rescaling
max_value = np.max(img_gray_copy)
print("Max value of img_gray_copy: ", max_value)
min_value = np.min(img_gray_copy)
print("Min value of img_gray_copy: ", min_value)
# pixel normalization: Scales values of the pixels in 0-1 range.
# min-max scaling = (X - Xmin) / (Xmax - Xmin), where X is a pixel of an image data
img_gray_normal = (img_gray_copy - min_value) / (max_value - min_value)
fig = plt.figure(figsize=(12, 10))
fig.add_subplot(1,2,1)
plt.axis("off")
plt.imshow(img_gray_copy, cmap='gray')
plt.title('Original image')
fig.add_subplot(1,2,2)
plt.axis("off")
plt.imshow(img_gray_normal, cmap='gray')
plt.title('Normalization result')
plt.show()


img_gray_hist = cv2.calcHist([img_gray_copy], [0], None, [256], [0, 1.0])
img_gray_normal_hist = cv2.calcHist([img_gray_normal], [0], None, [256], [0, 1.0])
fig = plt.figure( figsize=(10, 8) ) # histograms
fig.add_subplot(1, 2, 1)
plt.plot(img_gray_hist)
plt.title('Original image')
plt.xticks(np.arange(0, 256, step=50), ['0', '0.2', '0.4', '0.6', '0.8', '1.0'])
fig.add_subplot(1, 2, 2)
plt.plot(img_gray_normal_hist)
plt.title('Normalization result')
plt.xticks(np.arange(0, 256, step=50), ['0', '0.2', '0.4', '0.6', '0.8', '1.0'])
plt.show()
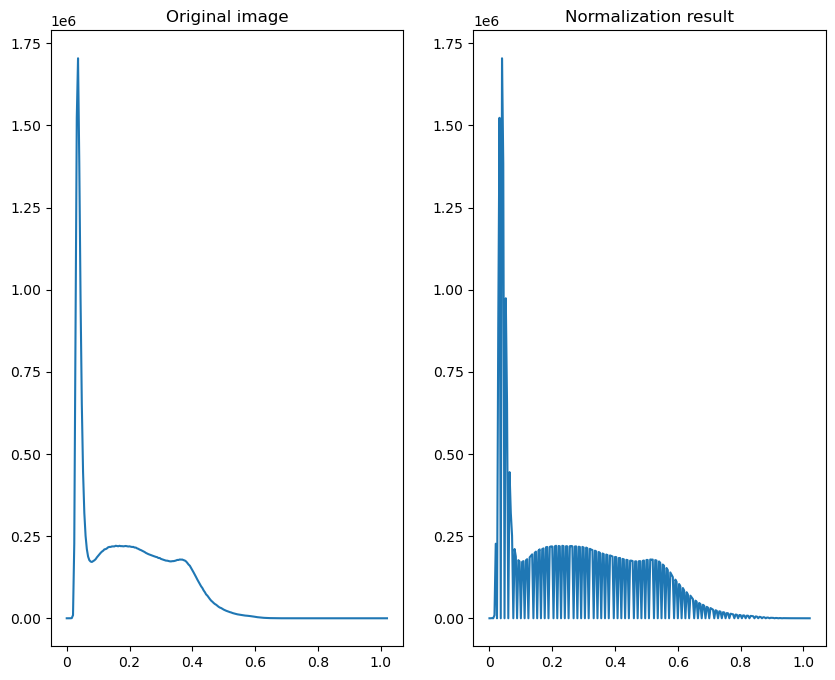
위 결과이미지 우측의 Rescale된 이미지는 좀 더 밝게 표현되며, 그 밑의 histogram은 그 이유를
잘 보여주고 있습니다. 좌측 대상 이미지의 histogram에서 알 수 있듯이, 픽셀값의 분포는
대략 0에서 0.4에 이르는 낮은 픽셀값 좁은 영역의 데이터분포를 갖지만 우측의 histogram은 0 - 0.8의
상대적으로 넓은 영역에 분포되어 있으므로 normalization된 이미지는 상대적으로 밝으며 좀 더 상세한
부분까지 표현가능한 것입니다.
이와같이 대상 이미지 데이터가 좁은영역에 픽셀값이 분포한 경우 normalization은 이미지 품질 개선의
효과를 보여줍니다.
Standardization
앞의 normalization과 병행되는 또다른 이미지 품질 개선 테크닉을 소개합니다.
대상 이미지의 픽셀값 분포가 넓은 영역에 고르지 않고 어둡거나 밝은 영역에 편향적으로 분포되어
있다면 - 예를들어 histogram에서 어두운 영역에 급격한 높은 봉우리를 갖고 밝은영역에 긴 꼬리를
갖을경우 - standardization이 큰 도움이 될것입니다.
이것은 픽셀값의 분포를 평균(mean) 0, 표준편차(standard deviation) 1 을 갖도록 scale시켜 줍니다.
즉, 픽셀값 분포의 대부분은 "0" 을 중심으로 위치함으로써 분포를 평탄하게 해줍니다.
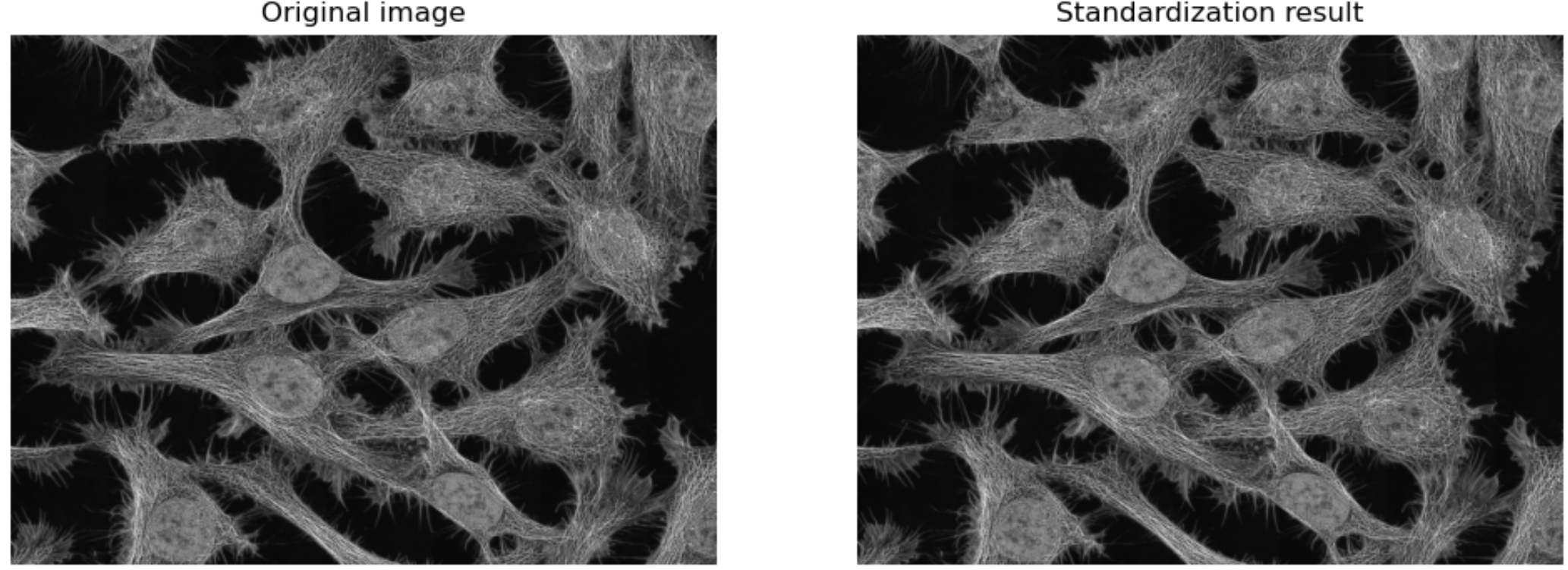
Standardization은 픽셀값에서 픽셀의 평균값을 뺀후 픽셀의 표준편차로 나누어 구합니다.
standardization = (X - Xmean) / Xstd, 여기서 X: 픽셀값, Xmean: 픽셀의 평균, Xstd: 픽셀의 표준편차
주의할점은 입력 이미지 데이터는 소수점 형식 (float32 등)의 변수로 변환해야 하며
각 픽셀별로 나눗셈 연산을 수행하므로 연산 결과값 또한 소수점 형식의 변수에 저장되어야 합니다.
# data type conversion and rescaling
img_gray_copy = img_gray.copy().astype(np.float32) # convert from uint8 to float32 type data
img_gray_copy /= 255. # rescaling
mean_value = np.mean(img_gray_copy);
print("Mean value of img_gray_copy: ", mean_value)
std_value = np.std(img_gray_copy);
print("Standard deviation of img_gray_copy: ", std_value)
# pixel Standardization:
# Scales values of the pixels to have 0 mean and unit (1) variance.
# standardization = (X - Xmean) / Xstd
img_gray_stand = (img_gray_copy - mean_value) / std_value
fig = plt.figure(figsize=(12, 10)) # (12,10)
fig.add_subplot(1, 2, 1)
plt.axis("off")
plt.imshow(img_gray_copy, cmap='gray')
plt.title('Original image')
fig.add_subplot(1, 2, 2)
plt.axis("off")
plt.imshow(img_gray_stand, cmap='gray')
plt.title('Standardization result')
plt.show()


img_gray_hist = cv2.calcHist([img_gray_copy], [0], None, [256], [0, 1.0])
img_gray_stand_hist = cv2.calcHist([img_gray_stand], [0], None, [256], [-2.0, 4.0])
fig = plt.figure(figsize=(10,8)) # histograms
fig.add_subplot(1, 2, 1)
plt.plot(img_gray_hist)
plt.title('Original image')
plt.xticks(np.arange(0, 256, step=50), ['0', '0.2', '0.4', '0.6', '0.8', '1.0'])
fig.add_subplot(1,2,2)
plt.plot(img_gray_stand_hist)
plt.title('Standardization result')
plt.xticks(np.arange(0, 256, step=50),['-2.0', '-1.0', '0', '1.0', '2.0', '3.0'])
plt.show()
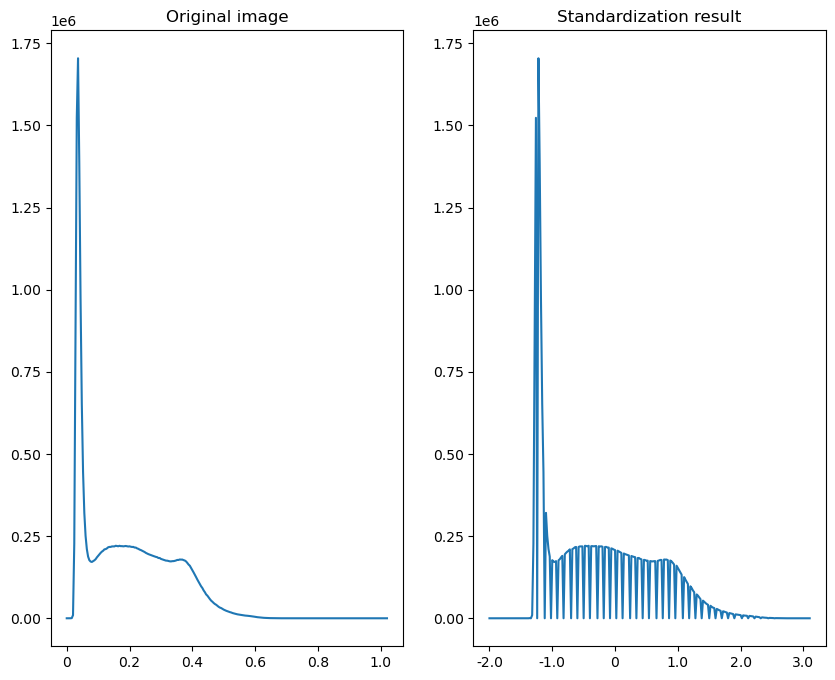
위의 결과 이미지 중 우측 이미지는 standardization을 수행한 결과로써 화질 개선의 결과는
유의미해 보이지 않지만, 밑의 histogram은 화질 개선의 결과를 잘 보여주고 있습니다.
우측의 histogram에서 픽셀값 분포는 -1.0에서 1.0 사이의 넓은 영역에 분포되어 있습니다.
만일, 0 이하의 픽셀들의 가중치를 낮추고 0을 초과하는 픽셀값에 가중치를 높게 연산을 한다면
이미지상에 뚜렷한 윤곽선 혹은 강조하고 싶은 영역 등에 하이라이트를 줄 수 있습니다.
또한 0을 초과하는 픽셀값만을 취하여 이진화가 가능하므로 대상 이미지에 여러 cell(세포)들이
혼재한 경우 특정 cell(세포)을 추출 혹은 필터 마스크를 수행할 시, 개인적으로 미세한 형태적
특징을 고려해 binarization 보다는 standardization을 선호 합니다.
출처1: https://unsplash.com/photos/jdfn7Z03Qa4
사진 작가: National Cancer Institute, Unsplash
Cells from cervical cancer – Splash에서 National Cancer Institute의 이 사진 다운로드
unsplash.com
'Python 영상처리 기초 with OpenCV' 카테고리의 다른 글
| [Python 영상처리 기초 7] 이미지의 화질개선 Contrast-Brightness-Gamma - part1 (2) | 2023.09.16 |
|---|---|
| [Python 영상처리 기초 6] 선형 공간 필터링 - Average, Laplacian, Sobel filters (0) | 2023.08.08 |
| [Python 영상처리 기초 4] 이미지의 픽셀값 다루기 - Crop, 선형연산 (2) | 2023.07.31 |
| [Python 영상처리 기초 3] 이미지의 픽셀값 다루기 - Gray scale, Histogram, Binarization (0) | 2023.07.29 |
| [Python 영상처리 기초 2] BGR컬러 이미지의 색상채널분리 - cv2.split( ), cv2.merge( ) (0) | 2023.07.28 |